Introducción a la Programación con R Tidyverse
Paul Efren Santos Andrade
Enero, 2021
Aprenderemos
Conceptos básicos de usando el IDE RStudio (Entorno de desarrollo integrado).
Escritura de codigos legibles y reproducibles.
Conceptos basicos de ciencia de datos.
Programas
Es necesario tener instalados:
Una versión reciente de R (~4.0.3), disponile de forma gratuita en cran.r-project.org
Una versión reciente de RStudio IDE (~1.2.5033), adisponible en www.rstudio.com/download.
Paquetes o librerias
Instalación
install.packages("vegan")install.packages("here")install.packages("readxl")- Esta acción solo se realiza una vez por computador.
Activación
library("here")library("tidyverse")library("vegan")- Siempre que se inicie una sesión en RStudio
Paquetes necesarios:
- Será necesario que instales los siguientes paquetes:
packages <- c("tidyverse", "janitor", "here", "writexl", "readxl") install.packages(packages) El lenguaje de programación.
El lenguaje de programación.
 Un programa para escribir código.
Un programa para escribir código.
El lenguaje de programación.
El lenguaje de programación.
- Valores (atomicos):
"cusco "## [1] "cusco "" Cusco"## [1] " Cusco""16-08-2021"## [1] "16-08-2021"1## [1] 11.2## [1] 1.2El lenguaje de programación.
- Creación de objetos:
- El operador usado para crear objetos es
<-
x <- 1x## [1] 1y <- "ser"y## [1] "ser"El lenguaje de programación.
- Creación de objetos:
- Para almacenar más de un elemento en un objeto se tiene el operador
c( ):
x <- c(1, 2, 3, 4, 5)x## [1] 1 2 3 4 5x <- c("El ", "dia", "cusco", "r", "programacion") x## [1] "El " "dia" "cusco" "r" "programacion"x <- c(1, 3, 4, 6, "ser")x## [1] "1" "3" "4" "6" "ser"El lenguaje de programación.
- Operadores
a. Secuencia ::
1:6## [1] 1 2 3 4 5 6b. Indexación []:
letters## [1] "a" "b" "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s"## [20] "t" "u" "v" "w" "x" "y" "z"letters[1:5]## [1] "a" "b" "c" "d" "e"letters[2]## [1] "b"letters[c(2, 4)]## [1] "b" "d"El lenguaje de programación.
- Operadores logicos
| Operador | Meaning |
|---|---|
| x < y | menor que |
| x > y | mayor que |
| x == y | igual |
| x <= y | menor igual que |
| x >= y | mayor igual que |
| x != y | diferente |
| x %in% y | pertenece al grupo |
El lenguaje de programación.
- Operadores boleanos
a. & - y
b. | - o
c. ! - negación
d. %in% - perteneciente a un grupo
El lenguaje de programación.
- Estructuras básicas:
- Vectores:
x <- c(1:6)x## [1] 1 2 3 4 5 6class(x)## [1] "integer"x <- c(1.5:5.5)x## [1] 1.5 2.5 3.5 4.5 5.5class(x)## [1] "numeric"El lenguaje de programación.
- Estructuras básicas:
- Vectores:
x <- c("a", "e", "i", "o", "u")x## [1] "a" "e" "i" "o" "u"class(x)## [1] "character"x <- c(TRUE, TRUE, FALSE, TRUE, FALSE)x## [1] TRUE TRUE FALSE TRUE FALSEclass(x)## [1] "logical"El lenguaje de programación.
- Estructuras básicas:
- Matrices:
matrix(1:10, ncol = 5)## [,1] [,2] [,3] [,4] [,5]## [1,] 1 3 5 7 9## [2,] 2 4 6 8 10matrix(letters[1:15], nrow = 3)## [,1] [,2] [,3] [,4] [,5]## [1,] "a" "d" "g" "j" "m" ## [2,] "b" "e" "h" "k" "n" ## [3,] "c" "f" "i" "l" "o"El lenguaje de programación.
- Estructuras básicas:
3.1 Dataframe
## mpg cyl disp hp drat wt qsec vs am gear carb## Mazda RX4 21.0 6 160.0 110 3.90 2.620 16.46 0 1 4 4## Mazda RX4 Wag 21.0 6 160.0 110 3.90 2.875 17.02 0 1 4 4## Datsun 710 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1## Hornet 4 Drive 21.4 6 258.0 110 3.08 3.215 19.44 1 0 3 1## Hornet Sportabout 18.7 8 360.0 175 3.15 3.440 17.02 0 0 3 2## Valiant 18.1 6 225.0 105 2.76 3.460 20.22 1 0 3 1## Duster 360 14.3 8 360.0 245 3.21 3.570 15.84 0 0 3 4## Merc 240D 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2## Merc 230 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2## Merc 280 19.2 6 167.6 123 3.92 3.440 18.30 1 0 4 4## Merc 280C 17.8 6 167.6 123 3.92 3.440 18.90 1 0 4 4## Merc 450SE 16.4 8 275.8 180 3.07 4.070 17.40 0 0 3 3## Merc 450SL 17.3 8 275.8 180 3.07 3.730 17.60 0 0 3 3## Merc 450SLC 15.2 8 275.8 180 3.07 3.780 18.00 0 0 3 3## Cadillac Fleetwood 10.4 8 472.0 205 2.93 5.250 17.98 0 0 3 4## Lincoln Continental 10.4 8 460.0 215 3.00 5.424 17.82 0 0 3 4## Chrysler Imperial 14.7 8 440.0 230 3.23 5.345 17.42 0 0 3 4## Fiat 128 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1## Honda Civic 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2## Toyota Corolla 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1## Toyota Corona 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1## Dodge Challenger 15.5 8 318.0 150 2.76 3.520 16.87 0 0 3 2## AMC Javelin 15.2 8 304.0 150 3.15 3.435 17.30 0 0 3 2## Camaro Z28 13.3 8 350.0 245 3.73 3.840 15.41 0 0 3 4## Pontiac Firebird 19.2 8 400.0 175 3.08 3.845 17.05 0 0 3 2## Fiat X1-9 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1## Porsche 914-2 26.0 4 120.3 91 4.43 2.140 16.70 0 1 5 2## Lotus Europa 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2## Ford Pantera L 15.8 8 351.0 264 4.22 3.170 14.50 0 1 5 4## Ferrari Dino 19.7 6 145.0 175 3.62 2.770 15.50 0 1 5 6## Maserati Bora 15.0 8 301.0 335 3.54 3.570 14.60 0 1 5 8## Volvo 142E 21.4 4 121.0 109 4.11 2.780 18.60 1 1 4 2El lenguaje de programación.
- Estructuras básicas:
3.2 Tibble
## # A tibble: 32 x 11## mpg cyl disp hp drat wt qsec vs am gear carb## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 21 6 160 110 3.9 2.62 16.5 0 1 4 4## 2 21 6 160 110 3.9 2.88 17.0 0 1 4 4## 3 22.8 4 108 93 3.85 2.32 18.6 1 1 4 1## 4 21.4 6 258 110 3.08 3.22 19.4 1 0 3 1## 5 18.7 8 360 175 3.15 3.44 17.0 0 0 3 2## 6 18.1 6 225 105 2.76 3.46 20.2 1 0 3 1## 7 14.3 8 360 245 3.21 3.57 15.8 0 0 3 4## 8 24.4 4 147. 62 3.69 3.19 20 1 0 4 2## 9 22.8 4 141. 95 3.92 3.15 22.9 1 0 4 2## 10 19.2 6 168. 123 3.92 3.44 18.3 1 0 4 4## # ... with 22 more rowsEl lenguaje de programación.
- Estructuras básicas:
- Listas:
list(x = 1:5, y = letters[5:8], programa = c("tidyr", "dplyr", "janitor"))## $x## [1] 1 2 3 4 5## ## $y## [1] "e" "f" "g" "h"## ## $programa## [1] "tidyr" "dplyr" "janitor"El lenguaje de programación.
- Estructuras básicas:
- Listas:
list(x = 1:5, matriz = matrix(1:6, ncol = 3))## $x## [1] 1 2 3 4 5## ## $matriz## [,1] [,2] [,3]## [1,] 1 3 5## [2,] 2 4 6El lenguaje de programación.
- Estructuras básicas:
- Listas:
list(matriz = matrix(LETTERS[c(1, 3, 5, 6, 3, 7, 1, 2, 21)], ncol = 3), mtcars = head(mtcars[1:4]))## $matriz## [,1] [,2] [,3]## [1,] "A" "F" "A" ## [2,] "C" "C" "B" ## [3,] "E" "G" "U" ## ## $mtcars## mpg cyl disp hp## Mazda RX4 21.0 6 160 110## Mazda RX4 Wag 21.0 6 160 110## Datsun 710 22.8 4 108 93## Hornet 4 Drive 21.4 6 258 110## Hornet Sportabout 18.7 8 360 175## Valiant 18.1 6 225 105El lenguaje de programación.
- Estructuras básicas:
- Listas:
list(x = 1:4, list(x = LETTERS[2:6], y = "ser"))## $x## [1] 1 2 3 4## ## [[2]]## [[2]]$x## [1] "B" "C" "D" "E" "F"## ## [[2]]$y## [1] "ser"El lenguaje de programación.
Funciones:
round(x, digits = 3)round: nombre de la función( ): complemeto de la funciónx ; digits = 3: argumentos de la función
c(1.234:3.234)## [1] 1.234 2.234 3.234round(c(1.234:3.234), digits = 2)## [1] 1.23 2.23 3.23El lenguaje de programación.
- Funciones:
Para tener ayuda respecto a una función:
?roundhelp(mean)Preguntas
Importar datos
Importar datos

library("readr")read_csv(".../")read_tsv(".../")read_delim(".../")
library(readxl)#' .xlsx - .xlsread_excel(".../")#' .xlsread_xls(".../")#' .xlsxread_xlsx(".../")Importar datos
- .csv
- Base:
anp <- read.csv("data/areas_naturales_peru.csv")- Tidyverse
anp <- read_csv("data/areas_naturales_peru.csv")Importar datos
- Excel
read_excel()
library(readxl)anp <- read_excel("data/areas_naturales_peru.xlsx")read_xlsx()
anp <- read_xlsx("data/areas_naturales_peru.xlsx")Importar datos
- .txt
data.table::fread()
library(data.table)anp <- fread(here("data", "areas_naturales_peru.txt"), skip = 2 )read.delim()
anp <- read.delim(here("data", "areas_naturales_peru.txt"))Importar datos
anp <- read_excel(here("data", "areas_naturales_peru.xlsx"))anp## # A tibble: 268 x 9## ...1 ...2 ...3 CREACIÓN ...5 MODIFICACIÓN ...7 ...8 ...9 ## <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>## 1 CATEGOR~ CÓDIGO CATEGO~ BASE LEGAL FECHA~ BASE LEGAL FECHA~ UBICACI~ EXTE~## 2 PARQUES~ PN 01 de Cut~ LEY Nº 13~ 08.09~ LEY Nº 28860 03.08~ CAJAMAR~ 8214~## 3 PARQUES~ PN 02 de Tin~ LEY Nº 15~ 14.05~ <NA> <NA> HUANUCO 4777 ## 4 PARQUES~ PN 03 del Ma~ D.S. Nº 6~ 29.05~ D.S. Nº 045-~ 11.07~ CUSCO y~ 1716~## 5 PARQUES~ PN 04 Huasca~ D.S. Nº 0~ 01.07~ <NA> <NA> ANCASH 3400~## 6 PARQUES~ PN 05 Cerros~ D.S. Nº 0~ 22.07~ D.S. Nº 046-~ 07.07~ TUMBES ~ 1517~## 7 PARQUES~ PN 06 del Rí~ D.S. Nº 0~ 11.08~ <NA> <NA> SAN MAR~ 2745~## 8 PARQUES~ PN 07 Yanach~ D.S. Nº 0~ 29.08~ <NA> <NA> PASCO 1220~## 9 PARQUES~ PN 08 Bahuaj~ D.S. Nº 0~ 17.07~ D.S. Nº 048-~ 04.09~ MADRE D~ 1091~## 10 PARQUES~ PN 09 Cordil~ D.S. Nº 0~ 21.05~ <NA> <NA> SAN MAR~ 1353~## # ... with 258 more rowsImportar datos
read_excel(file, skip = 1)
anp <- read_excel(here("data", "areas_naturales_peru.xlsx"), skip = 1)anp## # A tibble: 267 x 9## CATEGORIAS...1 CÓDIGO CATEGORIAS...3 `BASE LEGAL...4` `FECHA PROMULGA~## <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES NACIO~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 ## 2 PARQUES NACIO~ PN 02 de Tingo María LEY Nº 15574 14.05.1965 ## 3 PARQUES NACIO~ PN 03 del Manu D.S. Nº 644-197~ 29.05.1973 ## 4 PARQUES NACIO~ PN 04 Huascarán D.S. Nº 0622-19~ 01.07.1975 ## 5 PARQUES NACIO~ PN 05 Cerros de Amo~ D.S. Nº 0800-19~ 22.07.1975 ## 6 PARQUES NACIO~ PN 06 del Río Abiseo D.S. Nº 064-198~ 11.08.1983 ## 7 PARQUES NACIO~ PN 07 Yanachaga - C~ D.S. Nº 068-198~ 29.08.1986 ## 8 PARQUES NACIO~ PN 08 Bahuaja - Son~ D.S. Nº 012-199~ 17.07.1996 ## 9 PARQUES NACIO~ PN 09 Cordillera Az~ D.S. Nº 031-200~ 21.05.2001 ## 10 PARQUES NACIO~ PN 10 Otishi D.S. Nº 003-200~ 14.01.2003 ## # ... with 257 more rows, and 4 more variables: `BASE LEGAL...6` <chr>, `FECHA## # PROMULGACIÓN...7` <chr>, `UBICACIÓN POLITICA` <chr>, `EXTENSION (ha)` <dbl>Importar datos
- Formato base
anp <- read_excel("data/areas_naturales_peru.xlsx"), skip = 1)anp- Usando Here
anp <- read_excel(here("data", "areas_naturales_peru.xlsx"), skip = 1)anp- Ambos formatos son equivales
Preguntas
Revisión de los datos
Revisión de los datos
- Las variables
names():
names(anp)## [1] "CATEGORIAS...1" "CÓDIGO" "CATEGORIAS...3" ## [4] "BASE LEGAL...4" "FECHA PROMULGACIÓN...5" "BASE LEGAL...6" ## [7] "FECHA PROMULGACIÓN...7" "UBICACIÓN POLITICA" "EXTENSION (ha)"Revisión de los datos
- Estructura
str():
str(anp)## tibble [267 x 9] (S3: tbl_df/tbl/data.frame)## $ CATEGORIAS...1 : chr [1:267] "PARQUES NACIONALES (15)" "PARQUES NACIONALES (15)" "PARQUES NACIONALES (15)" "PARQUES NACIONALES (15)" ...## $ CÓDIGO : chr [1:267] "PN 01" "PN 02" "PN 03" "PN 04" ...## $ CATEGORIAS...3 : chr [1:267] "de Cutervo" "de Tingo María" "del Manu" "Huascarán" ...## $ BASE LEGAL...4 : chr [1:267] "LEY Nº 13694" "LEY Nº 15574" "D.S. Nº 644-1973-AG" "D.S. Nº 0622-1975-AG" ...## $ FECHA PROMULGACIÓN...5: chr [1:267] "08.09.1961" "14.05.1965" "29.05.1973" "01.07.1975" ...## $ BASE LEGAL...6 : chr [1:267] "LEY Nº 28860" NA "D.S. Nº 045-2002-AG" NA ...## $ FECHA PROMULGACIÓN...7: chr [1:267] "03.08.2006" NA "11.07.2002" NA ...## $ UBICACIÓN POLITICA : chr [1:267] "CAJAMARCA" "HUANUCO" "CUSCO y MADRE DE DIOS" "ANCASH" ...## $ EXTENSION (ha) : num [1:267] 8214 4777 1716295 340000 151767 ...Revisión de los datos
- Estructura
glimpse():
glimpse(anp)## Rows: 267## Columns: 9## $ CATEGORIAS...1 <chr> "PARQUES NACIONALES (15)", "PARQUE...## $ CÓDIGO <chr> "PN 01", "PN 02", "PN 03", "PN 04", "PN 05...## $ CATEGORIAS...3 <chr> "de Cutervo", "de Tingo María", "del Manu"...## $ `BASE LEGAL...4` <chr> "LEY Nº 13694", "LEY Nº 15574", "D.S. Nº 6...## $ `FECHA PROMULGACIÓN...5` <chr> "08.09.1961", "14.05.1965", "29.05.1973", ...## $ `BASE LEGAL...6` <chr> "LEY Nº 28860", NA, "D.S. Nº 045-2002-AG",...## $ `FECHA PROMULGACIÓN...7` <chr> "03.08.2006", NA, "11.07.2002", NA, "07.07...## $ `UBICACIÓN POLITICA` <chr> "CAJAMARCA", "HUANUCO", "CUSCO y MADRE DE ...## $ `EXTENSION (ha)` <dbl> 8214.23, 4777.00, 1716295.22, 340000.00, 1...Nombres de las variables
- Evitar caracteres especiales.
- No utilizar mayusculas.
- No dejar espacios entre las palabras.
Error
## [1] "CATEGORIAS...1" "CÓDIGO" "CATEGORIAS...3" ## [4] "BASE LEGAL...4" "FECHA PROMULGACIÓN...5" "BASE LEGAL...6" ## [7] "FECHA PROMULGACIÓN...7" "UBICACIÓN POLITICA" "EXTENSION (ha)"Correcto
## [1] "categorias_1" "codigo" "categorias_3" ## [4] "base_legal_4" "fecha_promulgacion_5" "base_legal_6" ## [7] "fecha_promulgacion_7" "ubicacion_politica" "extension_ha"Limpieza de los nombres
library(janitor)clean_names(anp)## # A tibble: 267 x 9## categorias_1 codigo categorias_3 base_legal_4 fecha_promulgac~ base_legal_6## <chr> <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES NAC~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 LEY Nº 28860## 2 PARQUES NAC~ PN 02 de Tingo Ma~ LEY Nº 15574 14.05.1965 <NA> ## 3 PARQUES NAC~ PN 03 del Manu D.S. Nº 644~ 29.05.1973 D.S. Nº 045~## 4 PARQUES NAC~ PN 04 Huascarán D.S. Nº 062~ 01.07.1975 <NA> ## 5 PARQUES NAC~ PN 05 Cerros de A~ D.S. Nº 080~ 22.07.1975 D.S. Nº 046~## 6 PARQUES NAC~ PN 06 del Río Abi~ D.S. Nº 064~ 11.08.1983 <NA> ## 7 PARQUES NAC~ PN 07 Yanachaga -~ D.S. Nº 068~ 29.08.1986 <NA> ## 8 PARQUES NAC~ PN 08 Bahuaja - S~ D.S. Nº 012~ 17.07.1996 D.S. Nº 048~## 9 PARQUES NAC~ PN 09 Cordillera ~ D.S. Nº 031~ 21.05.2001 <NA> ## 10 PARQUES NAC~ PN 10 Otishi D.S. Nº 003~ 14.01.2003 D.S. Nº 021~## # ... with 257 more rows, and 3 more variables: fecha_promulgacion_7 <chr>,## # ubicacion_politica <chr>, extension_ha <dbl>Limpieza de los nombres
- Periodos:
## [1] "ser.ser.ser"- Mayusculas:
## [1] "PesoDeSemilla"- Camelcase:
## [1] "peso_de_semilla"## [1] "altura_parcela"Revisión de los datos
install.packages("visdat")library(visdat)visdat::vis_dat(anp)Revisión de los datos
Pipe

pipe
anp %>% head()## # A tibble: 6 x 9## CATEGORIAS...1 CÓDIGO CATEGORIAS...3 `BASE LEGAL...4` `FECHA PROMULGA~## <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES NACIO~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 ## 2 PARQUES NACIO~ PN 02 de Tingo María LEY Nº 15574 14.05.1965 ## 3 PARQUES NACIO~ PN 03 del Manu D.S. Nº 644-197~ 29.05.1973 ## 4 PARQUES NACIO~ PN 04 Huascarán D.S. Nº 0622-19~ 01.07.1975 ## 5 PARQUES NACIO~ PN 05 Cerros de Amo~ D.S. Nº 0800-19~ 22.07.1975 ## 6 PARQUES NACIO~ PN 06 del Río Abiseo D.S. Nº 064-198~ 11.08.1983 ## # ... with 4 more variables: `BASE LEGAL...6` <chr>, `FECHA## # PROMULGACIÓN...7` <chr>, `UBICACIÓN POLITICA` <chr>, `EXTENSION (ha)` <dbl>- Shortcut:
Ctrl + Shift + M
pipe Ctrl + Shift + M
anp %>% clean_names()## # A tibble: 267 x 9## categorias_1 codigo categorias_3 base_legal_4 fecha_promulgac~ base_legal_6## <chr> <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES NAC~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 LEY Nº 28860## 2 PARQUES NAC~ PN 02 de Tingo Ma~ LEY Nº 15574 14.05.1965 <NA> ## 3 PARQUES NAC~ PN 03 del Manu D.S. Nº 644~ 29.05.1973 D.S. Nº 045~## 4 PARQUES NAC~ PN 04 Huascarán D.S. Nº 062~ 01.07.1975 <NA> ## 5 PARQUES NAC~ PN 05 Cerros de A~ D.S. Nº 080~ 22.07.1975 D.S. Nº 046~## 6 PARQUES NAC~ PN 06 del Río Abi~ D.S. Nº 064~ 11.08.1983 <NA> ## 7 PARQUES NAC~ PN 07 Yanachaga -~ D.S. Nº 068~ 29.08.1986 <NA> ## 8 PARQUES NAC~ PN 08 Bahuaja - S~ D.S. Nº 012~ 17.07.1996 D.S. Nº 048~## 9 PARQUES NAC~ PN 09 Cordillera ~ D.S. Nº 031~ 21.05.2001 <NA> ## 10 PARQUES NAC~ PN 10 Otishi D.S. Nº 003~ 14.01.2003 D.S. Nº 021~## # ... with 257 more rows, and 3 more variables: fecha_promulgacion_7 <chr>,## # ubicacion_politica <chr>, extension_ha <dbl>Pipe %>%
anp <- read_excel(here("data", "areas_naturales_peru.xlsx"), skip = 1) %>% clean_names()anp## # A tibble: 267 x 9## categorias_1 codigo categorias_3 base_legal_4 fecha_promulgac~ base_legal_6## <chr> <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES NAC~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 LEY Nº 28860## 2 PARQUES NAC~ PN 02 de Tingo Ma~ LEY Nº 15574 14.05.1965 <NA> ## 3 PARQUES NAC~ PN 03 del Manu D.S. Nº 644~ 29.05.1973 D.S. Nº 045~## 4 PARQUES NAC~ PN 04 Huascarán D.S. Nº 062~ 01.07.1975 <NA> ## 5 PARQUES NAC~ PN 05 Cerros de A~ D.S. Nº 080~ 22.07.1975 D.S. Nº 046~## 6 PARQUES NAC~ PN 06 del Río Abi~ D.S. Nº 064~ 11.08.1983 <NA> ## 7 PARQUES NAC~ PN 07 Yanachaga -~ D.S. Nº 068~ 29.08.1986 <NA> ## 8 PARQUES NAC~ PN 08 Bahuaja - S~ D.S. Nº 012~ 17.07.1996 D.S. Nº 048~## 9 PARQUES NAC~ PN 09 Cordillera ~ D.S. Nº 031~ 21.05.2001 <NA> ## 10 PARQUES NAC~ PN 10 Otishi D.S. Nº 003~ 14.01.2003 D.S. Nº 021~## # ... with 257 more rows, and 3 more variables: fecha_promulgacion_7 <chr>,## # ubicacion_politica <chr>, extension_ha <dbl>Pipe %>%
anp %>% vis_dat()Renombrar
anp1 <- read_excel(here("data", "areas_naturales_peru.xlsx"), skip = 0) %>% clean_names()- Datos crudos de excel
## # A tibble: 2 x 9## x1 x2 x3 creacion x5 modificacion x7 x8 x9 ## <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> ## 1 CATEGORIAS CÓDIGO CATEG~ BASE LEG~ FECHA ~ BASE LEGAL FECHA ~ UBICAC~ EXTEN~## 2 PARQUES N~ PN 01 de Cu~ LEY Nº 1~ 08.09.~ LEY Nº 28860 03.08.~ CAJAMA~ 8214.~- Nombres de las columnas
## [1] "x1" "x2" "x3" "creacion" "x5" ## [6] "modificacion" "x7" "x8" "x9"Renombrar
anp <- read_excel(here("data", "areas_naturales_peru.xlsx"), skip = 1) %>% clean_names()- Omitiendo la columna que contiene metadata
## # A tibble: 3 x 9## categorias_1 codigo categorias_3 base_legal_4 fecha_promulgac~ base_legal_6## <chr> <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES NAC~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 LEY Nº 28860## 2 PARQUES NAC~ PN 02 de Tingo Ma~ LEY Nº 15574 14.05.1965 <NA> ## 3 PARQUES NAC~ PN 03 del Manu D.S. Nº 644~ 29.05.1973 D.S. Nº 045~## # ... with 3 more variables: fecha_promulgacion_7 <chr>,## # ubicacion_politica <chr>, extension_ha <dbl>- Nombres de las columnas
## [1] "categorias_1" "codigo" "categorias_3" ## [4] "base_legal_4" "fecha_promulgacion_5" "base_legal_6" ## [7] "fecha_promulgacion_7" "ubicacion_politica" "extension_ha"Renombrar
colnames()
## [1] "x1" "x2" "x3" "creacion" "x5" ## [6] "modificacion" "x7" "x8" "x9"## [1] "categorias_1" "codigo" "categorias_3" ## [4] "base_legal_4" "fecha_promulgacion_5" "base_legal_6" ## [7] "fecha_promulgacion_7" "ubicacion_politica" "extension_ha"- Cambiar los nombres :
correct_names <- c( "categoria", "codigo", "nombre_area", "base_legal_creacion", "fecha_promulgacion_creacion", "base_legal_modificacion", "fecha_promulgacion__modificacion", "ubicacion_politica", "extension_ha" )Renombrar
colnames()
colnames(anp) <- correct_namesanp %>% head(n = 3)## # A tibble: 3 x 9## categoria codigo nombre_area base_legal_crea~ fecha_promulgac~## <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES ~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 ## 2 PARQUES ~ PN 02 de Tingo M~ LEY Nº 15574 14.05.1965 ## 3 PARQUES ~ PN 03 del Manu D.S. Nº 644-197~ 29.05.1973 ## # ... with 4 more variables: base_legal_modificacion <chr>,## # fecha_promulgacion__modificacion <chr>, ubicacion_politica <chr>,## # extension_ha <dbl>names(anp)## [1] "categoria" "codigo" ## [3] "nombre_area" "base_legal_creacion" ## [5] "fecha_promulgacion_creacion" "base_legal_modificacion" ## [7] "fecha_promulgacion__modificacion" "ubicacion_politica" ## [9] "extension_ha"Renombrar
set_names()
anp_2 <- anp %>% set_names(correct_names)anp_2## # A tibble: 267 x 9## categoria codigo nombre_area base_legal_crea~ fecha_promulgac~## <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES ~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 ## 2 PARQUES ~ PN 02 de Tingo M~ LEY Nº 15574 14.05.1965 ## 3 PARQUES ~ PN 03 del Manu D.S. Nº 644-197~ 29.05.1973 ## 4 PARQUES ~ PN 04 Huascarán D.S. Nº 0622-19~ 01.07.1975 ## 5 PARQUES ~ PN 05 Cerros de ~ D.S. Nº 0800-19~ 22.07.1975 ## 6 PARQUES ~ PN 06 del Río Ab~ D.S. Nº 064-198~ 11.08.1983 ## 7 PARQUES ~ PN 07 Yanachaga ~ D.S. Nº 068-198~ 29.08.1986 ## 8 PARQUES ~ PN 08 Bahuaja - ~ D.S. Nº 012-199~ 17.07.1996 ## 9 PARQUES ~ PN 09 Cordillera~ D.S. Nº 031-200~ 21.05.2001 ## 10 PARQUES ~ PN 10 Otishi D.S. Nº 003-200~ 14.01.2003 ## # ... with 257 more rows, and 4 more variables: base_legal_modificacion <chr>,## # fecha_promulgacion__modificacion <chr>, ubicacion_politica <chr>,## # extension_ha <dbl>names(anp_2)## [1] "categoria" "codigo" ## [3] "nombre_area" "base_legal_creacion" ## [5] "fecha_promulgacion_creacion" "base_legal_modificacion" ## [7] "fecha_promulgacion__modificacion" "ubicacion_politica" ## [9] "extension_ha"Preguntas
dplyr

dplyr - verbs
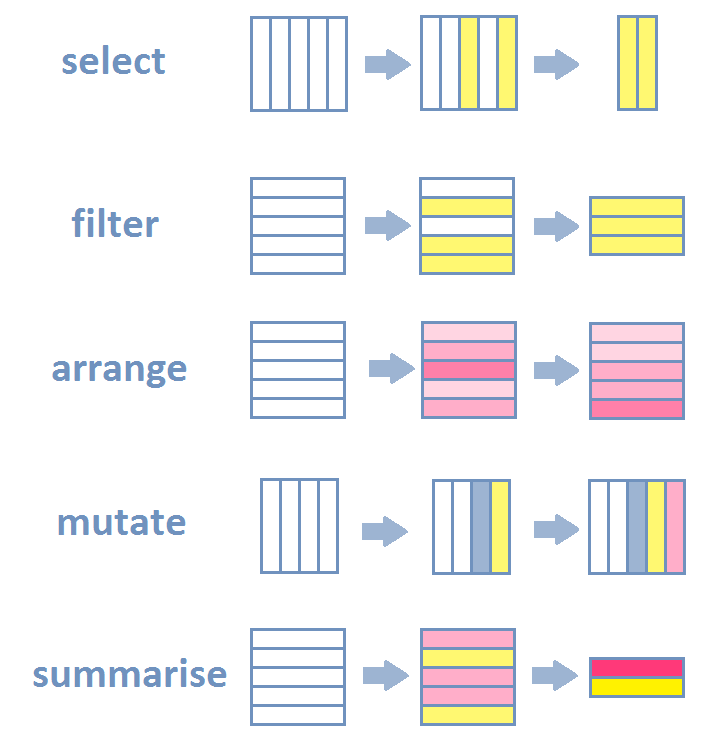
select() Actúa sobre columnas:
- Selección por nombre
select(anp, nombre_area)## # A tibble: 267 x 1## nombre_area ## <chr> ## 1 de Cutervo ## 2 de Tingo María ## 3 del Manu ## 4 Huascarán ## 5 Cerros de Amotape ## 6 del Río Abiseo ## 7 Yanachaga - Chemillén## 8 Bahuaja - Sonene ## 9 Cordillera Azul ## 10 Otishi ## # ... with 257 more rows- Usando
%>%
anp %>% select(nombre_area)## # A tibble: 267 x 1## nombre_area ## <chr> ## 1 de Cutervo ## 2 de Tingo María ## 3 del Manu ## 4 Huascarán ## 5 Cerros de Amotape ## 6 del Río Abiseo ## 7 Yanachaga - Chemillén## 8 Bahuaja - Sonene ## 9 Cordillera Azul ## 10 Otishi ## # ... with 257 more rowsselect(): df %>% select(variables)
- Selección numérica
anp %>% select(1)## # A tibble: 267 x 1## categoria ## <chr> ## 1 PARQUES NACIONALES (15)## 2 PARQUES NACIONALES (15)## 3 PARQUES NACIONALES (15)## 4 PARQUES NACIONALES (15)## 5 PARQUES NACIONALES (15)## 6 PARQUES NACIONALES (15)## 7 PARQUES NACIONALES (15)## 8 PARQUES NACIONALES (15)## 9 PARQUES NACIONALES (15)## 10 PARQUES NACIONALES (15)## # ... with 257 more rowsselect(): df %>% select(variables)
- Selección por intervalos
anp %>% select(base_legal_creacion:fecha_promulgacion__modificacion)## # A tibble: 267 x 4## base_legal_creac~ fecha_promulgacion~ base_legal_modific~ fecha_promulgacion~## <chr> <chr> <chr> <chr> ## 1 LEY Nº 13694 08.09.1961 LEY Nº 28860 03.08.2006 ## 2 LEY Nº 15574 14.05.1965 <NA> <NA> ## 3 D.S. Nº 644-1973~ 29.05.1973 D.S. Nº 045-2002-AG 11.07.2002 ## 4 D.S. Nº 0622-197~ 01.07.1975 <NA> <NA> ## 5 D.S. Nº 0800-197~ 22.07.1975 D.S. Nº 046-2006-A~ 07.07.2006 / 11.11~## 6 D.S. Nº 064-1983~ 11.08.1983 <NA> <NA> ## 7 D.S. Nº 068-1986~ 29.08.1986 <NA> <NA> ## 8 D.S. Nº 012-1996~ 17.07.1996 D.S. Nº 048-2000-AG 04.09.2000 ## 9 D.S. Nº 031-2001~ 21.05.2001 <NA> <NA> ## 10 D.S. Nº 003-2003~ 14.01.2003 D.S. Nº 021-2003-AG 30.05.2003 ## # ... with 257 more rowsselect(): df %>% select(variables)
- Selección por intervalos
anp %>% select(4:9)## # A tibble: 267 x 6## base_legal_crea~ fecha_promulgac~ base_legal_modi~ fecha_promulgac~## <chr> <chr> <chr> <chr> ## 1 LEY Nº 13694 08.09.1961 LEY Nº 28860 03.08.2006 ## 2 LEY Nº 15574 14.05.1965 <NA> <NA> ## 3 D.S. Nº 644-197~ 29.05.1973 D.S. Nº 045-200~ 11.07.2002 ## 4 D.S. Nº 0622-19~ 01.07.1975 <NA> <NA> ## 5 D.S. Nº 0800-19~ 22.07.1975 D.S. Nº 046-200~ 07.07.2006 / 11~## 6 D.S. Nº 064-198~ 11.08.1983 <NA> <NA> ## 7 D.S. Nº 068-198~ 29.08.1986 <NA> <NA> ## 8 D.S. Nº 012-199~ 17.07.1996 D.S. Nº 048-200~ 04.09.2000 ## 9 D.S. Nº 031-200~ 21.05.2001 <NA> <NA> ## 10 D.S. Nº 003-200~ 14.01.2003 D.S. Nº 021-200~ 30.05.2003 ## # ... with 257 more rows, and 2 more variables: ubicacion_politica <chr>,## # extension_ha <dbl>select(): starts_with()
df %>% select(starts_with("texto"))
anp %>% select(starts_with("base"))## # A tibble: 267 x 2## base_legal_creacion base_legal_modificacion ## <chr> <chr> ## 1 LEY Nº 13694 LEY Nº 28860 ## 2 LEY Nº 15574 <NA> ## 3 D.S. Nº 644-1973-AG D.S. Nº 045-2002-AG ## 4 D.S. Nº 0622-1975-AG <NA> ## 5 D.S. Nº 0800-1975-AG D.S. Nº 046-2006-AG; Ley N°30359## 6 D.S. Nº 064-1983-AG <NA> ## 7 D.S. Nº 068-1986-AG <NA> ## 8 D.S. Nº 012-1996-AG D.S. Nº 048-2000-AG ## 9 D.S. Nº 031-2001-AG <NA> ## 10 D.S. Nº 003-2003-AG D.S. Nº 021-2003-AG ## # ... with 257 more rowsselect(): ends_with()
df %>% select(ends_with("texto"))
anp %>% select(ends_with("cion"))## # A tibble: 267 x 4## base_legal_creac~ fecha_promulgacion~ base_legal_modific~ fecha_promulgacion~## <chr> <chr> <chr> <chr> ## 1 LEY Nº 13694 08.09.1961 LEY Nº 28860 03.08.2006 ## 2 LEY Nº 15574 14.05.1965 <NA> <NA> ## 3 D.S. Nº 644-1973~ 29.05.1973 D.S. Nº 045-2002-AG 11.07.2002 ## 4 D.S. Nº 0622-197~ 01.07.1975 <NA> <NA> ## 5 D.S. Nº 0800-197~ 22.07.1975 D.S. Nº 046-2006-A~ 07.07.2006 / 11.11~## 6 D.S. Nº 064-1983~ 11.08.1983 <NA> <NA> ## 7 D.S. Nº 068-1986~ 29.08.1986 <NA> <NA> ## 8 D.S. Nº 012-1996~ 17.07.1996 D.S. Nº 048-2000-AG 04.09.2000 ## 9 D.S. Nº 031-2001~ 21.05.2001 <NA> <NA> ## 10 D.S. Nº 003-2003~ 14.01.2003 D.S. Nº 021-2003-AG 30.05.2003 ## # ... with 257 more rowsselect(): contains()
df %>% select(contains("texto"))
anp %>% select(contains("legal"))## # A tibble: 267 x 2## base_legal_creacion base_legal_modificacion ## <chr> <chr> ## 1 LEY Nº 13694 LEY Nº 28860 ## 2 LEY Nº 15574 <NA> ## 3 D.S. Nº 644-1973-AG D.S. Nº 045-2002-AG ## 4 D.S. Nº 0622-1975-AG <NA> ## 5 D.S. Nº 0800-1975-AG D.S. Nº 046-2006-AG; Ley N°30359## 6 D.S. Nº 064-1983-AG <NA> ## 7 D.S. Nº 068-1986-AG <NA> ## 8 D.S. Nº 012-1996-AG D.S. Nº 048-2000-AG ## 9 D.S. Nº 031-2001-AG <NA> ## 10 D.S. Nº 003-2003-AG D.S. Nº 021-2003-AG ## # ... with 257 more rows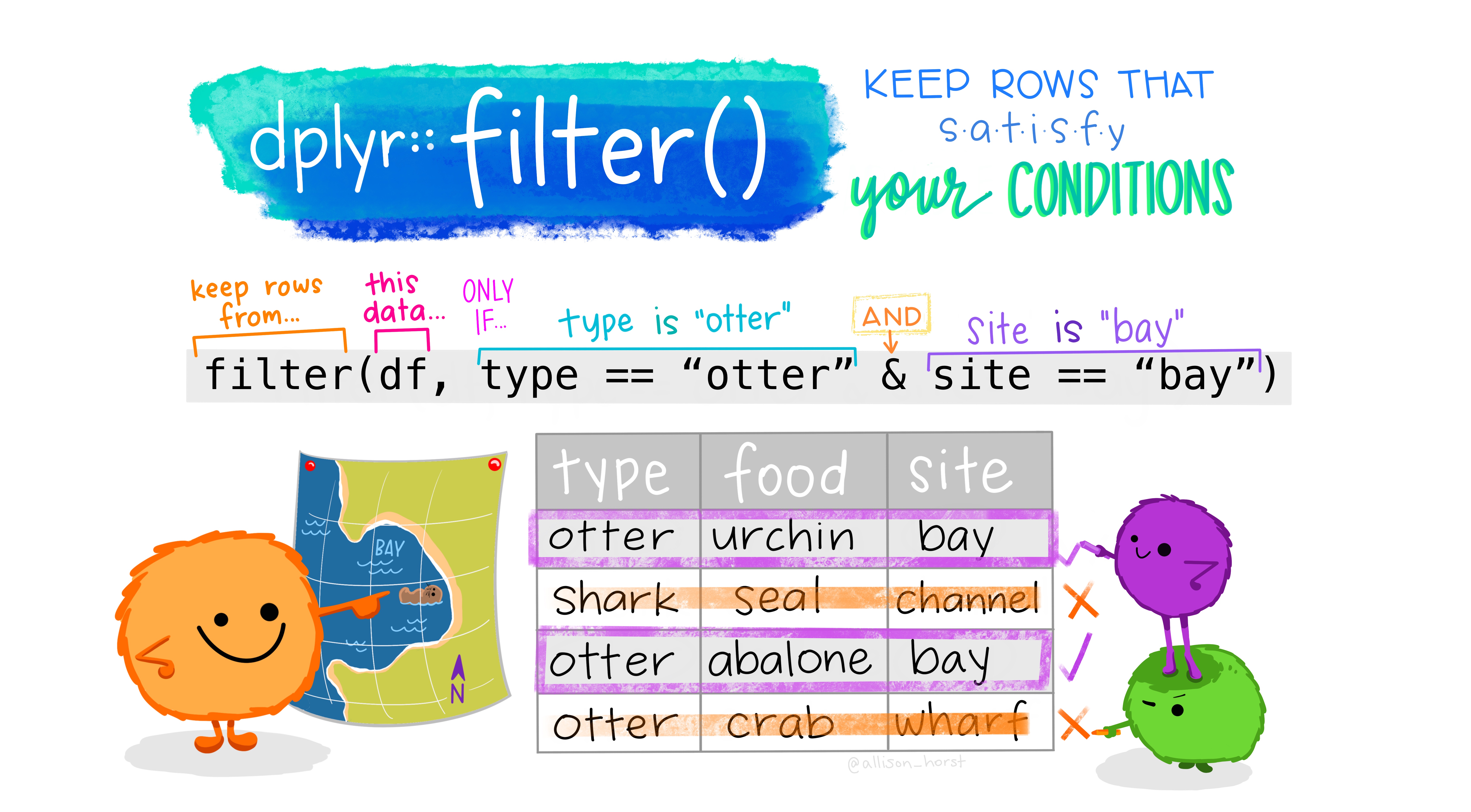
filter() Actúa sobre filas:
df %>% filter(condicion)
anp %>% filter(codigo == "PN 01")## # A tibble: 1 x 9## categoria codigo nombre_area base_legal_crea~ fecha_promulgac~## <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES ~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 ## # ... with 4 more variables: base_legal_modificacion <chr>,## # fecha_promulgacion__modificacion <chr>, ubicacion_politica <chr>,## # extension_ha <dbl>df %>% filter(condicion)
La condición está conformada por:
- Variable
var, nombre de la variable o columna. - Operador:
var == "x", valores devariguales a "x"var != "y", valores devardiferentes a "y"var == 1, valores devarigual a 1var == 1L, valores devarigual a 1var %in% c("a", "b", "c"), valores devarincluidos en el vectorc("a", "b", "c")!var %in% c("a", "b", "c"), valores devarno incluidos en el vectorc("a", "b", "c")
filter(varriable == vector)
anp %>% filter(nombre_area == "Huascarán")## # A tibble: 1 x 9## categoria codigo nombre_area base_legal_crea~ fecha_promulgac~## <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES ~ PN 04 Huascarán D.S. Nº 0622-19~ 01.07.1975 ## # ... with 4 more variables: base_legal_modificacion <chr>,## # fecha_promulgacion__modificacion <chr>, ubicacion_politica <chr>,## # extension_ha <dbl>filter(varriable %in% vector)
anp %>% filter(codigo %in% c("PN 01", "PN 02", "PN 03"))## # A tibble: 3 x 9## categoria codigo nombre_area base_legal_crea~ fecha_promulgac~## <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES ~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 ## 2 PARQUES ~ PN 02 de Tingo M~ LEY Nº 15574 14.05.1965 ## 3 PARQUES ~ PN 03 del Manu D.S. Nº 644-197~ 29.05.1973 ## # ... with 4 more variables: base_legal_modificacion <chr>,## # fecha_promulgacion__modificacion <chr>, ubicacion_politica <chr>,## # extension_ha <dbl>filter() ! variable - %in% vector:
anp %>% filter(!codigo %in% c("PN 01", "PN 02", "PN 03"))## # A tibble: 264 x 9## categoria codigo nombre_area base_legal_crea~ fecha_promulgac~## <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES ~ PN 04 Huascarán D.S. Nº 0622-19~ 01.07.1975 ## 2 PARQUES ~ PN 05 Cerros de ~ D.S. Nº 0800-19~ 22.07.1975 ## 3 PARQUES ~ PN 06 del Río Ab~ D.S. Nº 064-198~ 11.08.1983 ## 4 PARQUES ~ PN 07 Yanachaga ~ D.S. Nº 068-198~ 29.08.1986 ## 5 PARQUES ~ PN 08 Bahuaja - ~ D.S. Nº 012-199~ 17.07.1996 ## 6 PARQUES ~ PN 09 Cordillera~ D.S. Nº 031-200~ 21.05.2001 ## 7 PARQUES ~ PN 10 Otishi D.S. Nº 003-200~ 14.01.2003 ## 8 PARQUES ~ PN 11 Alto Purús D.S. Nº 040-200~ 18.11.2004 ## 9 PARQUES ~ PN 12 Ichigkat M~ D.S. Nº 023-200~ 09.08.2007 ## 10 PARQUES ~ PN 13 Güeppí-Sek~ D.S. Nº 006-201~ 25.10.2012 ## # ... with 254 more rows, and 4 more variables: base_legal_modificacion <chr>,## # fecha_promulgacion__modificacion <chr>, ubicacion_politica <chr>,## # extension_ha <dbl>filter - str_detect()
- Coincidencia parcial de caracteres
df %>% filter(str_detect(var, "texto"))
anp %>% filter(str_detect(nombre_area, "Alto")) %>% select(categoria, nombre_area)## # A tibble: 5 x 2## categoria nombre_area ## <chr> <chr> ## 1 PARQUES NACIONALES (15) Alto Purús ## 2 BOSQUES DE PROTECCION (6) Alto Mayo ## 3 AREAS DE CONSERVACION REGIONAL (25) Alto Nanay-Pintuyacu-Chambira ## 4 areas de conservacion privada (143) Abra Patricia - Alto Nieva ## 5 areas de conservacion privada (143) Bosque Seco de Chililique Altofilter()
- Filtros con más de una condición
anp %>% filter(str_detect(nombre_area, "Alto"), extension_ha > 1000) %>% select(categoria, nombre_area, extension_ha)## # A tibble: 4 x 3## categoria nombre_area extension_ha## <chr> <chr> <dbl>## 1 PARQUES NACIONALES (15) Alto Purús 2510694.## 2 BOSQUES DE PROTECCION (6) Alto Mayo 182000 ## 3 AREAS DE CONSERVACION REGIONAL (25) Alto Nanay-Pintuyacu-Chambira 954635.## 4 areas de conservacion privada (143) Abra Patricia - Alto Nieva 1416.filter()
Filtros con más de una condición
&:
anp %>% filter(str_detect(nombre_area, "Alto") & extension_ha > 1000) %>% select(categoria, nombre_area, extension_ha)## # A tibble: 4 x 3## categoria nombre_area extension_ha## <chr> <chr> <dbl>## 1 PARQUES NACIONALES (15) Alto Purús 2510694.## 2 BOSQUES DE PROTECCION (6) Alto Mayo 182000 ## 3 AREAS DE CONSERVACION REGIONAL (25) Alto Nanay-Pintuyacu-Chambira 954635.## 4 areas de conservacion privada (143) Abra Patricia - Alto Nieva 1416.filter()
|O
anp %>% filter(str_detect(nombre_area, "Alto") | ubicacion_politica == "CUSCO") %>% select(categoria, nombre_area, ubicacion_politica)## # A tibble: 29 x 3## categoria nombre_area ubicacion_politica ## <chr> <chr> <chr> ## 1 PARQUES NACIONALES ~ Alto Purús UCAYALI y MADRE DE ~## 2 SANTUARIOS NACIONALES (~ Megantoni CUSCO ## 3 SANTUARIOS HISTORICOS ~ de Machupicchu CUSCO ## 4 BOSQUES DE PROTECCION (6) Alto Mayo SAN MARTIN ## 5 AREAS DE CONSERVACION REG~ Choquequirao CUSCO ## 6 AREAS DE CONSERVACION REG~ Alto Nanay-Pintuyacu-Chambira LORETO ## 7 AREAS DE CONSERVACION REG~ Tres Cañones CUSCO ## 8 AREAS DE CONSERVACION REG~ Ausangate CUSCO ## 9 areas de conservacion pri~ Abra Patricia - Alto Nieva AMAZONAS ## 10 areas de conservacion pri~ Abra Málaga Thastayoc - Roya~ CUSCO ## # ... with 19 more rows
mutate()
- Esta función permite generar nuevas o modificar las variables existentes.
anp %>% select(nombre_area, extension_ha) %>% mutate(extencion_doble = extension_ha * 2) %>% head()## # A tibble: 6 x 3## nombre_area extension_ha extencion_doble## <chr> <dbl> <dbl>## 1 de Cutervo 8214. 16428.## 2 de Tingo María 4777 9554 ## 3 del Manu 1716295. 3432590.## 4 Huascarán 340000 680000 ## 5 Cerros de Amotape 151767. 303535.## 6 del Río Abiseo 274520 549040Operadores matemáticos básicos
+suma-resta*Multiplicación/División^Potencia
mutate() - str_trim()
- Elimina los espacios blancos al inicio y final de una cadena de texto.
x <- " primer curso "x## [1] " primer curso "str_trim(x)## [1] "primer curso"str_trim(" caso tipo")## [1] "caso tipo"x <- "peru"x == " peru"## [1] FALSEx == str_trim(" peru")## [1] TRUEmutate() - str_trim()
anp %>% select(nombre_area) %>% mutate(nchar1 = str_length(nombre_area), nchar2 = str_length(str_trim(nombre_area)), compara = (nchar1 == nchar2)) %>% filter(compara == FALSE)## # A tibble: 4 x 4## nombre_area nchar1 nchar2 compara## <chr> <int> <int> <lgl> ## 1 "\r\nNor Yauyos - Cochas" 21 19 FALSE ## 2 "\r\nAledaño a la Bocatoma del Canal Nuevo Imperial" 48 46 FALSE ## 3 "\r\nEl Angolo" 11 9 FALSE ## 4 "\r\nCordillera Escalera" 21 19 FALSE
mutate() - str_squish()
anp %>% mutate(nombre_area = str_squish(nombre_area))## # A tibble: 267 x 9## categoria codigo nombre_area base_legal_crea~ fecha_promulgac~## <chr> <chr> <chr> <chr> <chr> ## 1 PARQUES ~ PN 01 de Cutervo LEY Nº 13694 08.09.1961 ## 2 PARQUES ~ PN 02 de Tingo M~ LEY Nº 15574 14.05.1965 ## 3 PARQUES ~ PN 03 del Manu D.S. Nº 644-197~ 29.05.1973 ## 4 PARQUES ~ PN 04 Huascarán D.S. Nº 0622-19~ 01.07.1975 ## 5 PARQUES ~ PN 05 Cerros de ~ D.S. Nº 0800-19~ 22.07.1975 ## 6 PARQUES ~ PN 06 del Río Ab~ D.S. Nº 064-198~ 11.08.1983 ## 7 PARQUES ~ PN 07 Yanachaga ~ D.S. Nº 068-198~ 29.08.1986 ## 8 PARQUES ~ PN 08 Bahuaja - ~ D.S. Nº 012-199~ 17.07.1996 ## 9 PARQUES ~ PN 09 Cordillera~ D.S. Nº 031-200~ 21.05.2001 ## 10 PARQUES ~ PN 10 Otishi D.S. Nº 003-200~ 14.01.2003 ## # ... with 257 more rows, and 4 more variables: base_legal_modificacion <chr>,## # fecha_promulgacion__modificacion <chr>, ubicacion_politica <chr>,## # extension_ha <dbl>mutate() - str_to_lower()
anp %>% select(categoria) %>% mutate(categoria_minuscula = str_to_lower(categoria))## # A tibble: 267 x 2## categoria categoria_minuscula ## <chr> <chr> ## 1 PARQUES NACIONALES (15) parques nacionales (15)## 2 PARQUES NACIONALES (15) parques nacionales (15)## 3 PARQUES NACIONALES (15) parques nacionales (15)## 4 PARQUES NACIONALES (15) parques nacionales (15)## 5 PARQUES NACIONALES (15) parques nacionales (15)## 6 PARQUES NACIONALES (15) parques nacionales (15)## 7 PARQUES NACIONALES (15) parques nacionales (15)## 8 PARQUES NACIONALES (15) parques nacionales (15)## 9 PARQUES NACIONALES (15) parques nacionales (15)## 10 PARQUES NACIONALES (15) parques nacionales (15)## # ... with 257 more rowsmutate() - str_to_upper()
anp %>% select(nombre_area) %>% mutate(nombre_area_mayusculas = str_to_upper(nombre_area))## # A tibble: 267 x 2## nombre_area nombre_area_mayusculas## <chr> <chr> ## 1 de Cutervo DE CUTERVO ## 2 de Tingo María DE TINGO MARÍA ## 3 del Manu DEL MANU ## 4 Huascarán HUASCARÁN ## 5 Cerros de Amotape CERROS DE AMOTAPE ## 6 del Río Abiseo DEL RÍO ABISEO ## 7 Yanachaga - Chemillén YANACHAGA - CHEMILLÉN ## 8 Bahuaja - Sonene BAHUAJA - SONENE ## 9 Cordillera Azul CORDILLERA AZUL ## 10 Otishi OTISHI ## # ... with 257 more rowsmutate() - str_to_title()
anp %>% select(ubicacion_politica) %>% mutate(ubica_pol_2 = str_to_title(ubicacion_politica))## # A tibble: 267 x 2## ubicacion_politica ubica_pol_2 ## <chr> <chr> ## 1 CAJAMARCA Cajamarca ## 2 HUANUCO Huanuco ## 3 CUSCO y MADRE DE DIOS Cusco Y Madre De Dios ## 4 ANCASH Ancash ## 5 TUMBES y PIURA Tumbes Y Piura ## 6 SAN MARTIN San Martin ## 7 PASCO Pasco ## 8 MADRE DE DIOS y PUNO Madre De Dios Y Puno ## 9 SAN MARTIN, LORETO, UCAYALI y HUANUCO San Martin, Loreto, Ucayali Y Huanuco## 10 JUNIN y CUSCO Junin Y Cusco ## # ... with 257 more rowsmutate() - str_to_sentence()
anp %>% select(categoria) %>% mutate(categoria_2 = str_to_sentence(categoria))## # A tibble: 267 x 2## categoria categoria_2 ## <chr> <chr> ## 1 PARQUES NACIONALES (15) Parques nacionales (15)## 2 PARQUES NACIONALES (15) Parques nacionales (15)## 3 PARQUES NACIONALES (15) Parques nacionales (15)## 4 PARQUES NACIONALES (15) Parques nacionales (15)## 5 PARQUES NACIONALES (15) Parques nacionales (15)## 6 PARQUES NACIONALES (15) Parques nacionales (15)## 7 PARQUES NACIONALES (15) Parques nacionales (15)## 8 PARQUES NACIONALES (15) Parques nacionales (15)## 9 PARQUES NACIONALES (15) Parques nacionales (15)## 10 PARQUES NACIONALES (15) Parques nacionales (15)## # ... with 257 more rowstm
install.packages("tm")library(tm)mutate() - tm::removePunctuation()
anp %>% select(categoria ) %>% mutate(categoria_clean = removePunctuation(categoria))## # A tibble: 267 x 2## categoria categoria_clean ## <chr> <chr> ## 1 PARQUES NACIONALES (15) PARQUES NACIONALES 15## 2 PARQUES NACIONALES (15) PARQUES NACIONALES 15## 3 PARQUES NACIONALES (15) PARQUES NACIONALES 15## 4 PARQUES NACIONALES (15) PARQUES NACIONALES 15## 5 PARQUES NACIONALES (15) PARQUES NACIONALES 15## 6 PARQUES NACIONALES (15) PARQUES NACIONALES 15## 7 PARQUES NACIONALES (15) PARQUES NACIONALES 15## 8 PARQUES NACIONALES (15) PARQUES NACIONALES 15## 9 PARQUES NACIONALES (15) PARQUES NACIONALES 15## 10 PARQUES NACIONALES (15) PARQUES NACIONALES 15## # ... with 257 more rowsmutate() - tm::removeNumbers()
anp %>% select(categoria ) %>% mutate(categoria_clean = removeNumbers(categoria))## # A tibble: 267 x 2## categoria categoria_clean ## <chr> <chr> ## 1 PARQUES NACIONALES (15) PARQUES NACIONALES ()## 2 PARQUES NACIONALES (15) PARQUES NACIONALES ()## 3 PARQUES NACIONALES (15) PARQUES NACIONALES ()## 4 PARQUES NACIONALES (15) PARQUES NACIONALES ()## 5 PARQUES NACIONALES (15) PARQUES NACIONALES ()## 6 PARQUES NACIONALES (15) PARQUES NACIONALES ()## 7 PARQUES NACIONALES (15) PARQUES NACIONALES ()## 8 PARQUES NACIONALES (15) PARQUES NACIONALES ()## 9 PARQUES NACIONALES (15) PARQUES NACIONALES ()## 10 PARQUES NACIONALES (15) PARQUES NACIONALES ()## # ... with 257 more rowsmutate() - tm::removePunctuation()
anp %>% select(categoria ) %>% mutate(categoria_clean = removePunctuation(categoria) %>% removeNumbers())## # A tibble: 267 x 2## categoria categoria_clean ## <chr> <chr> ## 1 PARQUES NACIONALES (15) "PARQUES NACIONALES "## 2 PARQUES NACIONALES (15) "PARQUES NACIONALES "## 3 PARQUES NACIONALES (15) "PARQUES NACIONALES "## 4 PARQUES NACIONALES (15) "PARQUES NACIONALES "## 5 PARQUES NACIONALES (15) "PARQUES NACIONALES "## 6 PARQUES NACIONALES (15) "PARQUES NACIONALES "## 7 PARQUES NACIONALES (15) "PARQUES NACIONALES "## 8 PARQUES NACIONALES (15) "PARQUES NACIONALES "## 9 PARQUES NACIONALES (15) "PARQUES NACIONALES "## 10 PARQUES NACIONALES (15) "PARQUES NACIONALES "## # ... with 257 more rowsmutate()
anp %>% select(categoria ) %>% mutate(categoria_clean = removePunctuation(categoria) %>% removeNumbers() %>% str_squish())## # A tibble: 267 x 2## categoria categoria_clean ## <chr> <chr> ## 1 PARQUES NACIONALES (15) PARQUES NACIONALES## 2 PARQUES NACIONALES (15) PARQUES NACIONALES## 3 PARQUES NACIONALES (15) PARQUES NACIONALES## 4 PARQUES NACIONALES (15) PARQUES NACIONALES## 5 PARQUES NACIONALES (15) PARQUES NACIONALES## 6 PARQUES NACIONALES (15) PARQUES NACIONALES## 7 PARQUES NACIONALES (15) PARQUES NACIONALES## 8 PARQUES NACIONALES (15) PARQUES NACIONALES## 9 PARQUES NACIONALES (15) PARQUES NACIONALES## 10 PARQUES NACIONALES (15) PARQUES NACIONALES## # ... with 257 more rowsmutate()
anp %>% select(categoria) %>% mutate(categoria = removePunctuation(categoria) %>% removeNumbers() %>% str_squish() %>% str_to_title())## # A tibble: 267 x 1## categoria ## <chr> ## 1 Parques Nacionales## 2 Parques Nacionales## 3 Parques Nacionales## 4 Parques Nacionales## 5 Parques Nacionales## 6 Parques Nacionales## 7 Parques Nacionales## 8 Parques Nacionales## 9 Parques Nacionales## 10 Parques Nacionales## # ... with 257 more rowsmutate()
anp %>% select(categoria, codigo) %>% mutate(categoria = removePunctuation(categoria) %>% removeNumbers() %>% str_squish() %>% str_to_title()) %>% head()## # A tibble: 6 x 2## categoria codigo## <chr> <chr> ## 1 Parques Nacionales PN 01 ## 2 Parques Nacionales PN 02 ## 3 Parques Nacionales PN 03 ## 4 Parques Nacionales PN 04 ## 5 Parques Nacionales PN 05 ## 6 Parques Nacionales PN 06mutate()
anp %>% select(categoria, codigo) %>% mutate(categoria = removePunctuation(categoria) %>% removeNumbers() %>% str_squish() %>% str_to_title()) %>% distinct(codigo) %>% flatten_chr()## [1] "PN 01" "PN 02" "PN 03" "PN 04" ## [5] "PN 05" "PN 06" "PN 07" "PN 08" ## [9] "PN 09" "PN 10" "PN 11" "PN 12" ## [13] "PN 13" "PN 14" "PN 15" "\r\nSN 01" ## [17] "SN 02" "SN 03" "SN 04" "SN 05" ## [21] "SN 06" "SN 07" "SN 08" "SN 09" ## [25] "SH 01" "SH 02" "SH 03" "SH 04" ## [29] "RN 01" "RN 02" "RN 03" "RN 04" ## [33] "RN 05" "RN 06" "RN 07" "RN 08" ## [37] "RN 09" "RN 10" "RN 11" "RN 12" ## [41] "RN13.01" "RN13.02" "RN13.03" "RN13.04-05" ## [45] "RN13.06" "RN13.07" "RN13.08" "RN13.09" ## [49] "RN13.10" "RN13.11" "RN13.12" "RN13.13-15" ## [53] "RN13.16" "RN13.17-18" "RN13.19" "RN13.20" ## [57] "RN13.21-23" "RN13.24-26" "RN13.27" "RN13.28" ## [61] "RN13.29" "RN13.30" "RN13.31" "RN13.32" ## [65] "RN13.33" "RN14" "RN15" "RVS 01" ## [69] "RVS 02" "RVS 03" "RP\r\nRP 01" "RP 02" ## [73] "RC\r\nRC 01" "RC 02" "RC 03" "RC 04" ## [77] "RC 05" "RC 06" "RC 07" "RC 08" ## [81] "RC 09" "RC10" "BP\r\nBP 01" "BP 02" ## [85] "BP 03" "BP 04" "BP 05" "BP 06" ## [89] "CC\r\nCC 01" "CC 02" "ZR\r\n01" "3" ## [93] "5" "7" "11" "13" ## [97] "14" "15" "17" "ACR\r\nACR 01"## [101] "ACR 02" "ACR 03" "ACR 04" "ACR 05" ## [105] "ACR 06" "ACR 07" "ACR 08" "ACR 09" ## [109] "ACR 10" "ACR 11" "ACR 12" "ACR 13" ## [113] "ACR 14" "ACR 15" "ACR 16" "ACR 17" ## [117] "ACR 18" "ACR 19" "ACR 20" "ACR 21" ## [121] "ACR 22" "ACR 23" "ACR 24" "ACR 25" ## [125] "1" "2" "4" "6" ## [129] "9" "10" "18" "19" ## [133] "20" "21" "22" "23" ## [137] "24" "25" "26" "27" ## [141] "28" "29" "30" "31" ## [145] "32" "33" "34" "35" ## [149] "36" "37" "38" "39" ## [153] "40" "41" "42" "43" ## [157] "44" "45" "46" "47" ## [161] "48" "49" "50" "51" ## [165] "52" "53" "54" "55" ## [169] "56" "57" "58" "59" ## [173] "60" "61" "62" "63" ## [177] "64" "65" "66" "67" ## [181] "68" "69" "70" "71" ## [185] "72" "73" "74" "75" ## [189] "76" "77" "78" "79" ## [193] "80" "81" "82" "83" ## [197] "84" "85" "86" "87" ## [201] "88" "89" "90" "91" ## [205] "92" "93" "94" "95" ## [209] "96" "97" "98" "99" ## [213] "100" "101" "102" "103" ## [217] "104" "105" "106" "107" ## [221] "108" "109" "110" "111" ## [225] "112" "113" "114" "115" ## [229] "116" "117" "118" "119" ## [233] "120" "121" "122" "123" ## [237] "124" "125" "126" "127" ## [241] "128" "129" "130" "131" ## [245] "132" "133" "134" "135" ## [249] "136" "137" "138" "139" ## [253] "140" "141" "142" "143" ## [257] "144" "145" "146" "147" ## [261] "148" "149"stringr.plus

library(remotes)remotes::install_github("johncassil/stringr.plus")library(stringr.plus)mutate() - stringr.plus::str_extract_before()
anp %>% select(categoria, base_legal_creacion) %>% mutate(tipo_documento = str_extract_before(base_legal_creacion, "Nº") %>% str_squish())## # A tibble: 267 x 3## categoria base_legal_creacion tipo_documento## <chr> <chr> <chr> ## 1 PARQUES NACIONALES (15) LEY Nº 13694 LEY ## 2 PARQUES NACIONALES (15) LEY Nº 15574 LEY ## 3 PARQUES NACIONALES (15) D.S. Nº 644-1973-AG D.S. ## 4 PARQUES NACIONALES (15) D.S. Nº 0622-1975-AG D.S. ## 5 PARQUES NACIONALES (15) D.S. Nº 0800-1975-AG D.S. ## 6 PARQUES NACIONALES (15) D.S. Nº 064-1983-AG D.S. ## 7 PARQUES NACIONALES (15) D.S. Nº 068-1986-AG D.S. ## 8 PARQUES NACIONALES (15) D.S. Nº 012-1996-AG D.S. ## 9 PARQUES NACIONALES (15) D.S. Nº 031-2001-AG D.S. ## 10 PARQUES NACIONALES (15) D.S. Nº 003-2003-AG D.S. ## # ... with 257 more rowsmutate() - stringr.plus::str_extract_after()
anp %>% select(categoria, base_legal_creacion) %>% mutate(numero_documento = str_extract_after(base_legal_creacion, "Nº") %>% str_squish())## # A tibble: 267 x 3## categoria base_legal_creacion numero_documento## <chr> <chr> <chr> ## 1 PARQUES NACIONALES (15) LEY Nº 13694 13694 ## 2 PARQUES NACIONALES (15) LEY Nº 15574 15574 ## 3 PARQUES NACIONALES (15) D.S. Nº 644-1973-AG 644-1973-AG ## 4 PARQUES NACIONALES (15) D.S. Nº 0622-1975-AG 0622-1975-AG ## 5 PARQUES NACIONALES (15) D.S. Nº 0800-1975-AG 0800-1975-AG ## 6 PARQUES NACIONALES (15) D.S. Nº 064-1983-AG 064-1983-AG ## 7 PARQUES NACIONALES (15) D.S. Nº 068-1986-AG 068-1986-AG ## 8 PARQUES NACIONALES (15) D.S. Nº 012-1996-AG 012-1996-AG ## 9 PARQUES NACIONALES (15) D.S. Nº 031-2001-AG 031-2001-AG ## 10 PARQUES NACIONALES (15) D.S. Nº 003-2003-AG 003-2003-AG ## # ... with 257 more rowsAreas naturales limpia
anp %>% separate_rows(ubicacion_politica, sep = " y |\\, | Y ") %>% filter(!is.na(fecha_promulgacion__modificacion)) %>% separate_rows(fecha_promulgacion__modificacion, sep = " \\/ |\\;|\\-") %>% separate_rows(base_legal_modificacion, sep = "\\;|\\/") %>% mutate(categoria = str_squish(categoria) %>% removeNumbers() %>% removePunctuation() %>% str_trim() %>% str_to_title(), codigo = str_squish(codigo), codigo = sub("[A-Z]{1,}|[A-Z]{1,} [A-Z]{1,}", "", codigo) %>% str_squish(), tipo_doc_crea = str_extract_before(base_legal_creacion, "Nº"), num_doc_crea = str_extract_after(base_legal_creacion, "Nº"), tipo_doc_modif = str_extract_before(base_legal_modificacion, "Nº"), num_doc_modif = str_extract_after(base_legal_modificacion, "Nº"), fecha_promulgacion_creacion = str_squish(fecha_promulgacion_creacion) %>% str_replace_all("\\.", "-") %>% lubridate::as_date(format = "%d-%m-%Y"), fecha_promulgacion__modificacion = str_squish(fecha_promulgacion__modificacion)%>% str_replace_all("\\.", "-") %>% lubridate::as_date(format = "%d-%m-%Y") ) %>% fill(tipo_doc_crea, .direction = "down") %>% fill(num_doc_crea, .direction = "down") %>% mutate_if(is.character, ~str_squish(.)) %>% select(-c(base_legal_creacion, base_legal_modificacion))## # A tibble: 42 x 11## categoria codigo nombre_area fecha_promulgac~ fecha_promulgac~## <chr> <chr> <chr> <date> <date> ## 1 Parques ~ 01 de Cutervo 1961-09-08 2006-08-03 ## 2 Parques ~ 03 del Manu 1973-05-29 2002-07-11 ## 3 Parques ~ 03 del Manu 1973-05-29 2002-07-11 ## 4 Parques ~ 05 Cerros de ~ 1975-07-22 2006-07-07 ## 5 Parques ~ 05 Cerros de ~ 1975-07-22 2006-07-07 ## 6 Parques ~ 05 Cerros de ~ 1975-07-22 2015-11-11 ## 7 Parques ~ 05 Cerros de ~ 1975-07-22 2015-11-11 ## 8 Parques ~ 05 Cerros de ~ 1975-07-22 2006-07-07 ## 9 Parques ~ 05 Cerros de ~ 1975-07-22 2006-07-07 ## 10 Parques ~ 05 Cerros de ~ 1975-07-22 2015-11-11 ## # ... with 32 more rows, and 6 more variables: ubicacion_politica <chr>,## # extension_ha <dbl>, tipo_doc_crea <chr>, num_doc_crea <chr>,## # tipo_doc_modif <chr>, num_doc_modif <chr>Preguntas
summarise() - summarize()
Genera nuevas bases de datos, partiendo de la base de datos original.
anp_clean %>% summarise(numero_categorias = n_distinct(categoria))## # A tibble: 1 x 1## numero_categorias## <int>## 1 12summarise() - summarize()
anp_clean %>% summarise(area_total = sum(extension_ha))## # A tibble: 1 x 1## area_total## <dbl>## 1 42756297.group_by()
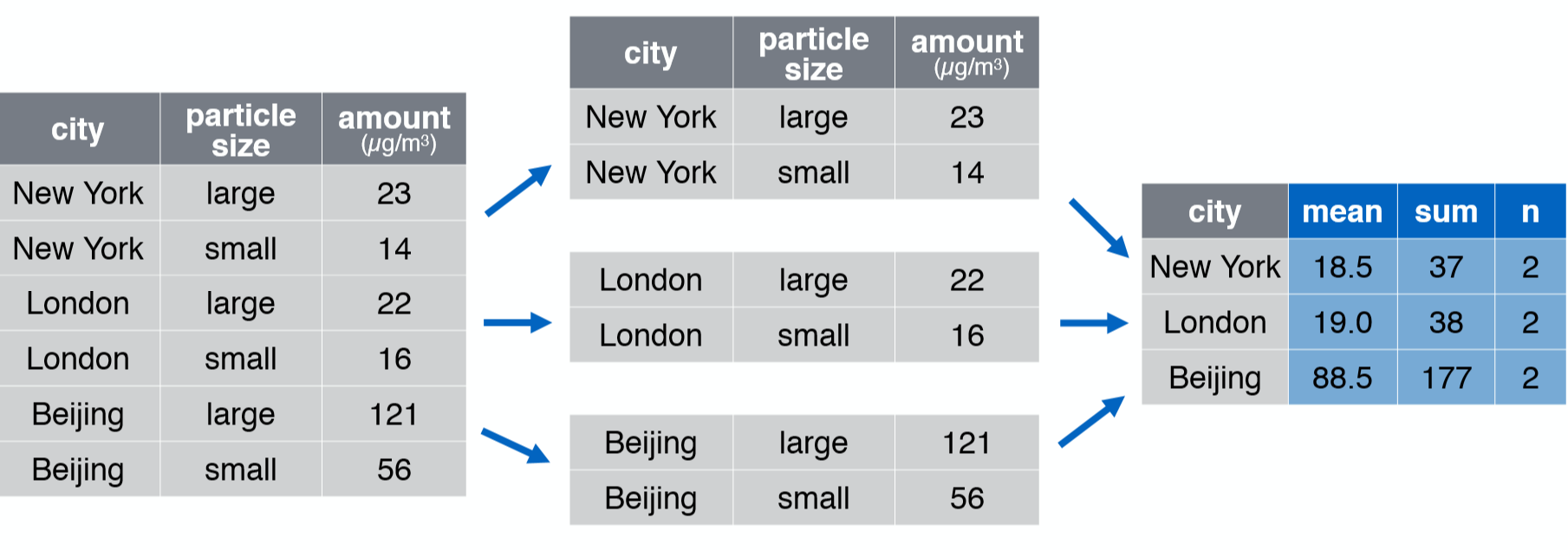
group_by()
anp_clean %>% group_by(categoria) %>% summarise(numero_areas = n_distinct(nombre_area))## # A tibble: 12 x 2## categoria numero_areas## * <chr> <int>## 1 Areas De Conservacion Privada 143## 2 Areas De Conservacion Regional 25## 3 Bosques De Proteccion 6## 4 Cotos De Caza 2## 5 Parques Nacionales 15## 6 Refugio De Vida Silvestre 3## 7 Reservas Comunales 10## 8 Reservas Nacionales 39## 9 Reservas Paisajísticas 2## 10 Santuarios Historicos 4## 11 Santuarios Nacionales 9## 12 Zonas Reservadas 9arrange() - desc()
anp_clean %>% group_by(categoria) %>% summarise(numero_areas = n_distinct(nombre_area)) %>% arrange(numero_areas)## # A tibble: 12 x 2## categoria numero_areas## <chr> <int>## 1 Cotos De Caza 2## 2 Reservas Paisajísticas 2## 3 Refugio De Vida Silvestre 3## 4 Santuarios Historicos 4## 5 Bosques De Proteccion 6## 6 Santuarios Nacionales 9## 7 Zonas Reservadas 9## 8 Reservas Comunales 10## 9 Parques Nacionales 15## 10 Areas De Conservacion Regional 25## 11 Reservas Nacionales 39## 12 Areas De Conservacion Privada 143arrange() - desc()
anp_clean %>% group_by(categoria) %>% summarise(numero_areas = n_distinct(nombre_area)) %>% arrange(desc(numero_areas))## # A tibble: 12 x 2## categoria numero_areas## <chr> <int>## 1 Areas De Conservacion Privada 143## 2 Reservas Nacionales 39## 3 Areas De Conservacion Regional 25## 4 Parques Nacionales 15## 5 Reservas Comunales 10## 6 Santuarios Nacionales 9## 7 Zonas Reservadas 9## 8 Bosques De Proteccion 6## 9 Santuarios Historicos 4## 10 Refugio De Vida Silvestre 3## 11 Cotos De Caza 2## 12 Reservas Paisajísticas 2Tidy Data

Tidy Data
tidyr

tidyr
library(vegan)data("BCI")BCI[1:10, 1:3]## Abarema.macradenia Vachellia.melanoceras Acalypha.diversifolia## 1 0 0 0## 2 0 0 0## 3 0 0 0## 4 0 0 0## 5 0 0 0## 6 0 0 0## 7 0 0 0## 8 0 0 0## 9 0 0 0## 10 1 0 0- Ayuda
?
? BCItidyr - pivot_longer()
BCI %>% pivot_longer(everything(), names_to = "especies", values_to = "abundancia")## # A tibble: 11,250 x 2## especies abundancia## <chr> <int>## 1 Abarema.macradenia 0## 2 Vachellia.melanoceras 0## 3 Acalypha.diversifolia 0## 4 Acalypha.macrostachya 0## 5 Adelia.triloba 0## 6 Aegiphila.panamensis 0## 7 Alchornea.costaricensis 2## 8 Alchornea.latifolia 0## 9 Alibertia.edulis 0## 10 Allophylus.psilospermus 0## # ... with 11,240 more rowstidyr - pivot_longer()
bci_tbl <- BCI %>% as_tibble( rownames = "plot_n")bci_tbl## # A tibble: 50 x 226## plot_n Abarema.macrade~ Vachellia.melan~ Acalypha.divers~ Acalypha.macros~## <chr> <int> <int> <int> <int>## 1 1 0 0 0 0## 2 2 0 0 0 0## 3 3 0 0 0 0## 4 4 0 0 0 0## 5 5 0 0 0 0## 6 6 0 0 0 0## 7 7 0 0 0 0## 8 8 0 0 0 0## 9 9 0 0 0 0## 10 10 1 0 0 0## # ... with 40 more rows, and 221 more variables: Adelia.triloba <int>,## # Aegiphila.panamensis <int>, Alchornea.costaricensis <int>,## # Alchornea.latifolia <int>, Alibertia.edulis <int>,## # Allophylus.psilospermus <int>, Alseis.blackiana <int>,## # Amaioua.corymbosa <int>, Anacardium.excelsum <int>, Andira.inermis <int>,## # Annona.spraguei <int>, Apeiba.glabra <int>, Apeiba.tibourbou <int>,## # Aspidosperma.desmanthum <int>, Astrocaryum.standleyanum <int>,## # Astronium.graveolens <int>, Attalea.butyracea <int>,## # Banara.guianensis <int>, Beilschmiedia.pendula <int>,## # Brosimum.alicastrum <int>, Brosimum.guianense <int>,## # Calophyllum.longifolium <int>, Casearia.aculeata <int>,## # Casearia.arborea <int>, Casearia.commersoniana <int>,## # Casearia.guianensis <int>, Casearia.sylvestris <int>,## # Cassipourea.guianensis <int>, Cavanillesia.platanifolia <int>,## # Cecropia.insignis <int>, Cecropia.obtusifolia <int>, Cedrela.odorata <int>,## # Ceiba.pentandra <int>, Celtis.schippii <int>, Cespedesia.spathulata <int>,## # Chamguava.schippii <int>, Chimarrhis.parviflora <int>,## # Maclura.tinctoria <int>, Chrysochlamys.eclipes <int>,## # Chrysophyllum.argenteum <int>, Chrysophyllum.cainito <int>,## # Coccoloba.coronata <int>, Coccoloba.manzinellensis <int>,## # Colubrina.glandulosa <int>, Cordia.alliodora <int>, Cordia.bicolor <int>,## # Cordia.lasiocalyx <int>, Coussarea.curvigemma <int>,## # Croton.billbergianus <int>, Cupania.cinerea <int>, Cupania.latifolia <int>,## # Cupania.rufescens <int>, Cupania.seemannii <int>,## # Dendropanax.arboreus <int>, Desmopsis.panamensis <int>,## # Diospyros.artanthifolia <int>, Dipteryx.oleifera <int>,## # Drypetes.standleyi <int>, Elaeis.oleifera <int>,## # Enterolobium.schomburgkii <int>, Erythrina.costaricensis <int>,## # Erythroxylum.macrophyllum <int>, Eugenia.florida <int>,## # Eugenia.galalonensis <int>, Eugenia.nesiotica <int>,## # Eugenia.oerstediana <int>, Faramea.occidentalis <int>,## # Ficus.colubrinae <int>, Ficus.costaricana <int>, Ficus.insipida <int>,## # Ficus.maxima <int>, Ficus.obtusifolia <int>, Ficus.popenoei <int>,## # Ficus.tonduzii <int>, Ficus.trigonata <int>, Ficus.yoponensis <int>,## # Garcinia.intermedia <int>, Garcinia.madruno <int>, Genipa.americana <int>,## # Guapira.myrtiflora <int>, Guarea.fuzzy <int>, Guarea.grandifolia <int>,## # Guarea.guidonia <int>, Guatteria.dumetorum <int>, Guazuma.ulmifolia <int>,## # Guettarda.foliacea <int>, Gustavia.superba <int>,## # Hampea.appendiculata <int>, Hasseltia.floribunda <int>,## # Heisteria.acuminata <int>, Heisteria.concinna <int>,## # Hirtella.americana <int>, Hirtella.triandra <int>, Hura.crepitans <int>,## # Hieronyma.alchorneoides <int>, Inga.acuminata <int>, Inga.cocleensis <int>,## # Inga.goldmanii <int>, Inga.laurina <int>, Inga.semialata <int>, ...tidyr - pivot_longer()
bci_long <- bci_tbl %>% pivot_longer(-plot_n, names_to = "especies", values_to = "abundancia")bci_long## # A tibble: 11,250 x 3## plot_n especies abundancia## <chr> <chr> <int>## 1 1 Abarema.macradenia 0## 2 1 Vachellia.melanoceras 0## 3 1 Acalypha.diversifolia 0## 4 1 Acalypha.macrostachya 0## 5 1 Adelia.triloba 0## 6 1 Aegiphila.panamensis 0## 7 1 Alchornea.costaricensis 2## 8 1 Alchornea.latifolia 0## 9 1 Alibertia.edulis 0## 10 1 Allophylus.psilospermus 0## # ... with 11,240 more rowstidyr - pivot_longer()
bci_long_1 <- bci_tbl %>% pivot_longer(-plot_n, names_to = "especies", values_to = "abundancia") %>% filter(abundancia != 0)bci_long_1## # A tibble: 4,539 x 3## plot_n especies abundancia## <chr> <chr> <int>## 1 1 Alchornea.costaricensis 2## 2 1 Alseis.blackiana 25## 3 1 Annona.spraguei 1## 4 1 Apeiba.glabra 13## 5 1 Apeiba.tibourbou 2## 6 1 Astronium.graveolens 6## 7 1 Beilschmiedia.pendula 4## 8 1 Brosimum.alicastrum 5## 9 1 Casearia.arborea 1## 10 1 Casearia.sylvestris 2## # ... with 4,529 more rowstidyr - pivot_wider()
bci_long %>% pivot_wider(names_from = especies, values_from = abundancia)## # A tibble: 50 x 226## plot_n Abarema.macrade~ Vachellia.melan~ Acalypha.divers~ Acalypha.macros~## <chr> <int> <int> <int> <int>## 1 1 0 0 0 0## 2 2 0 0 0 0## 3 3 0 0 0 0## 4 4 0 0 0 0## 5 5 0 0 0 0## 6 6 0 0 0 0## 7 7 0 0 0 0## 8 8 0 0 0 0## 9 9 0 0 0 0## 10 10 1 0 0 0## # ... with 40 more rows, and 221 more variables: Adelia.triloba <int>,## # Aegiphila.panamensis <int>, Alchornea.costaricensis <int>,## # Alchornea.latifolia <int>, Alibertia.edulis <int>,## # Allophylus.psilospermus <int>, Alseis.blackiana <int>,## # Amaioua.corymbosa <int>, Anacardium.excelsum <int>, Andira.inermis <int>,## # Annona.spraguei <int>, Apeiba.glabra <int>, Apeiba.tibourbou <int>,## # Aspidosperma.desmanthum <int>, Astrocaryum.standleyanum <int>,## # Astronium.graveolens <int>, Attalea.butyracea <int>,## # Banara.guianensis <int>, Beilschmiedia.pendula <int>,## # Brosimum.alicastrum <int>, Brosimum.guianense <int>,## # Calophyllum.longifolium <int>, Casearia.aculeata <int>,## # Casearia.arborea <int>, Casearia.commersoniana <int>,## # Casearia.guianensis <int>, Casearia.sylvestris <int>,## # Cassipourea.guianensis <int>, Cavanillesia.platanifolia <int>,## # Cecropia.insignis <int>, Cecropia.obtusifolia <int>, Cedrela.odorata <int>,## # Ceiba.pentandra <int>, Celtis.schippii <int>, Cespedesia.spathulata <int>,## # Chamguava.schippii <int>, Chimarrhis.parviflora <int>,## # Maclura.tinctoria <int>, Chrysochlamys.eclipes <int>,## # Chrysophyllum.argenteum <int>, Chrysophyllum.cainito <int>,## # Coccoloba.coronata <int>, Coccoloba.manzinellensis <int>,## # Colubrina.glandulosa <int>, Cordia.alliodora <int>, Cordia.bicolor <int>,## # Cordia.lasiocalyx <int>, Coussarea.curvigemma <int>,## # Croton.billbergianus <int>, Cupania.cinerea <int>, Cupania.latifolia <int>,## # Cupania.rufescens <int>, Cupania.seemannii <int>,## # Dendropanax.arboreus <int>, Desmopsis.panamensis <int>,## # Diospyros.artanthifolia <int>, Dipteryx.oleifera <int>,## # Drypetes.standleyi <int>, Elaeis.oleifera <int>,## # Enterolobium.schomburgkii <int>, Erythrina.costaricensis <int>,## # Erythroxylum.macrophyllum <int>, Eugenia.florida <int>,## # Eugenia.galalonensis <int>, Eugenia.nesiotica <int>,## # Eugenia.oerstediana <int>, Faramea.occidentalis <int>,## # Ficus.colubrinae <int>, Ficus.costaricana <int>, Ficus.insipida <int>,## # Ficus.maxima <int>, Ficus.obtusifolia <int>, Ficus.popenoei <int>,## # Ficus.tonduzii <int>, Ficus.trigonata <int>, Ficus.yoponensis <int>,## # Garcinia.intermedia <int>, Garcinia.madruno <int>, Genipa.americana <int>,## # Guapira.myrtiflora <int>, Guarea.fuzzy <int>, Guarea.grandifolia <int>,## # Guarea.guidonia <int>, Guatteria.dumetorum <int>, Guazuma.ulmifolia <int>,## # Guettarda.foliacea <int>, Gustavia.superba <int>,## # Hampea.appendiculata <int>, Hasseltia.floribunda <int>,## # Heisteria.acuminata <int>, Heisteria.concinna <int>,## # Hirtella.americana <int>, Hirtella.triandra <int>, Hura.crepitans <int>,## # Hieronyma.alchorneoides <int>, Inga.acuminata <int>, Inga.cocleensis <int>,## # Inga.goldmanii <int>, Inga.laurina <int>, Inga.semialata <int>, ...tidyr - pivot_wider()
bci_long_1 %>% pivot_wider(names_from = especies, values_from = abundancia)## # A tibble: 50 x 226## plot_n Alchornea.costa~ Alseis.blackiana Annona.spraguei Apeiba.glabra## <chr> <int> <int> <int> <int>## 1 1 2 25 1 13## 2 2 1 26 NA 12## 3 3 2 18 1 6## 4 4 18 23 NA 3## 5 5 3 16 NA 4## 6 6 2 14 NA 10## 7 7 NA 18 NA 5## 8 8 2 14 1 4## 9 9 2 16 1 5## 10 10 2 14 NA 5## # ... with 40 more rows, and 221 more variables: Apeiba.tibourbou <int>,## # Astronium.graveolens <int>, Beilschmiedia.pendula <int>,## # Brosimum.alicastrum <int>, Casearia.arborea <int>,## # Casearia.sylvestris <int>, Cassipourea.guianensis <int>,## # Cecropia.insignis <int>, Chrysophyllum.argenteum <int>,## # Cordia.alliodora <int>, Cordia.bicolor <int>, Cordia.lasiocalyx <int>,## # Croton.billbergianus <int>, Cupania.seemannii <int>,## # Diospyros.artanthifolia <int>, Dipteryx.oleifera <int>,## # Drypetes.standleyi <int>, Eugenia.oerstediana <int>,## # Faramea.occidentalis <int>, Ficus.maxima <int>, Ficus.yoponensis <int>,## # Garcinia.madruno <int>, Guapira.myrtiflora <int>, Guarea.fuzzy <int>,## # Guarea.guidonia <int>, Guatteria.dumetorum <int>, Guettarda.foliacea <int>,## # Gustavia.superba <int>, Hasseltia.floribunda <int>,## # Heisteria.concinna <int>, Hirtella.triandra <int>, Inga.cocleensis <int>,## # Inga.punctata <int>, Inga.sapindoides <int>, Jacaranda.copaia <int>,## # Lacistema.aggregatum <int>, Lacmellea.panamensis <int>,## # Lonchocarpus.heptaphyllus <int>, Luehea.seemannii <int>,## # Maquira.guianensis.costaricana <int>, Marila.laxiflora <int>,## # Maytenus.schippii <int>, Miconia.argentea <int>, Mosannona.garwoodii <int>,## # Myrcia.gatunensis <int>, Nectandra.purpurea <int>,## # Ochroma.pyramidale <int>, Ocotea.whitei <int>, Oenocarpus.mapora <int>,## # Platymiscium.pinnatum <int>, Platypodium.elegans <int>,## # Poulsenia.armata <int>, Pourouma.bicolor <int>, Pouteria.reticulata <int>,## # Prioria.copaifera <int>, Protium.costaricense <int>,## # Protium.panamense <int>, Protium.tenuifolium <int>,## # Pterocarpus.rohrii <int>, Quararibea.asterolepis <int>,## # Randia.armata <int>, Simarouba.amara <int>, Siparuna.guianensis <int>,## # Sloanea.terniflora <int>, Socratea.exorrhiza <int>, Sorocea.affinis <int>,## # Spondias.mombin <int>, Spondias.radlkoferi <int>, Sterculia.apetala <int>,## # Swartzia.simplex.var.grandiflora <int>,## # Swartzia.simplex.continentalis <int>, Handroanthus.guayacan <int>,## # Tabebuia.rosea <int>, Tabernaemontana.arborea <int>,## # Tachigali.versicolor <int>, Talisia.princeps <int>,## # Terminalia.amazonia <int>, Tetragastris.panamensis <int>,## # Theobroma.cacao <int>, Trattinnickia.aspera <int>,## # Trichilia.tuberculata <int>, Trophis.caucana <int>, Trophis.racemosa <int>,## # Unonopsis.pittieri <int>, Virola.sebifera <int>, Virola.surinamensis <int>,## # Xylopia.macrantha <int>, Zanthoxylum.ekmanii <int>,## # Zanthoxylum.panamense <int>, Astrocaryum.standleyanum <int>,## # Attalea.butyracea <int>, Calophyllum.longifolium <int>,## # Ceiba.pentandra <int>, Dendropanax.arboreus <int>,## # Erythroxylum.macrophyllum <int>, Eugenia.florida <int>,## # Ficus.colubrinae <int>, Garcinia.intermedia <int>,## # Hieronyma.alchorneoides <int>, Inga.spectabilis <int>, ...tidyr - pivot_wider()
bci_long_1 %>% pivot_wider(names_from = especies, values_from = abundancia, values_fill = 0)## # A tibble: 50 x 226## plot_n Alchornea.costa~ Alseis.blackiana Annona.spraguei Apeiba.glabra## <chr> <int> <int> <int> <int>## 1 1 2 25 1 13## 2 2 1 26 0 12## 3 3 2 18 1 6## 4 4 18 23 0 3## 5 5 3 16 0 4## 6 6 2 14 0 10## 7 7 0 18 0 5## 8 8 2 14 1 4## 9 9 2 16 1 5## 10 10 2 14 0 5## # ... with 40 more rows, and 221 more variables: Apeiba.tibourbou <int>,## # Astronium.graveolens <int>, Beilschmiedia.pendula <int>,## # Brosimum.alicastrum <int>, Casearia.arborea <int>,## # Casearia.sylvestris <int>, Cassipourea.guianensis <int>,## # Cecropia.insignis <int>, Chrysophyllum.argenteum <int>,## # Cordia.alliodora <int>, Cordia.bicolor <int>, Cordia.lasiocalyx <int>,## # Croton.billbergianus <int>, Cupania.seemannii <int>,## # Diospyros.artanthifolia <int>, Dipteryx.oleifera <int>,## # Drypetes.standleyi <int>, Eugenia.oerstediana <int>,## # Faramea.occidentalis <int>, Ficus.maxima <int>, Ficus.yoponensis <int>,## # Garcinia.madruno <int>, Guapira.myrtiflora <int>, Guarea.fuzzy <int>,## # Guarea.guidonia <int>, Guatteria.dumetorum <int>, Guettarda.foliacea <int>,## # Gustavia.superba <int>, Hasseltia.floribunda <int>,## # Heisteria.concinna <int>, Hirtella.triandra <int>, Inga.cocleensis <int>,## # Inga.punctata <int>, Inga.sapindoides <int>, Jacaranda.copaia <int>,## # Lacistema.aggregatum <int>, Lacmellea.panamensis <int>,## # Lonchocarpus.heptaphyllus <int>, Luehea.seemannii <int>,## # Maquira.guianensis.costaricana <int>, Marila.laxiflora <int>,## # Maytenus.schippii <int>, Miconia.argentea <int>, Mosannona.garwoodii <int>,## # Myrcia.gatunensis <int>, Nectandra.purpurea <int>,## # Ochroma.pyramidale <int>, Ocotea.whitei <int>, Oenocarpus.mapora <int>,## # Platymiscium.pinnatum <int>, Platypodium.elegans <int>,## # Poulsenia.armata <int>, Pourouma.bicolor <int>, Pouteria.reticulata <int>,## # Prioria.copaifera <int>, Protium.costaricense <int>,## # Protium.panamense <int>, Protium.tenuifolium <int>,## # Pterocarpus.rohrii <int>, Quararibea.asterolepis <int>,## # Randia.armata <int>, Simarouba.amara <int>, Siparuna.guianensis <int>,## # Sloanea.terniflora <int>, Socratea.exorrhiza <int>, Sorocea.affinis <int>,## # Spondias.mombin <int>, Spondias.radlkoferi <int>, Sterculia.apetala <int>,## # Swartzia.simplex.var.grandiflora <int>,## # Swartzia.simplex.continentalis <int>, Handroanthus.guayacan <int>,## # Tabebuia.rosea <int>, Tabernaemontana.arborea <int>,## # Tachigali.versicolor <int>, Talisia.princeps <int>,## # Terminalia.amazonia <int>, Tetragastris.panamensis <int>,## # Theobroma.cacao <int>, Trattinnickia.aspera <int>,## # Trichilia.tuberculata <int>, Trophis.caucana <int>, Trophis.racemosa <int>,## # Unonopsis.pittieri <int>, Virola.sebifera <int>, Virola.surinamensis <int>,## # Xylopia.macrantha <int>, Zanthoxylum.ekmanii <int>,## # Zanthoxylum.panamense <int>, Astrocaryum.standleyanum <int>,## # Attalea.butyracea <int>, Calophyllum.longifolium <int>,## # Ceiba.pentandra <int>, Dendropanax.arboreus <int>,## # Erythroxylum.macrophyllum <int>, Eugenia.florida <int>,## # Ficus.colubrinae <int>, Garcinia.intermedia <int>,## # Hieronyma.alchorneoides <int>, Inga.spectabilis <int>, ...